JCIM|VenomPred2.0:基于AI的药物分子毒性预测工具
人工智能和机器学习方法在计算毒理学和药物设计中的应用越来越受欢迎,这一方法在评估化合物安全性、优化ADMET以及满足3R原则(the rules of 3R)等领域被认为非常具有前景。
2024年4月8日,来自比萨大学的研究人员在Journal of Chemical Information and Modeling上发表研究:VenomPred 2.0: A Novel In Silico Platform for an Extended and Human Interpretable Toxicological Profiling of Small Molecules。

介绍了基于机器学习的用于毒理预测工具VenomPred 2.0,它提供了多种毒性预测,包括致癌性、致突变性、肝毒性、极性口服毒性、皮肤刺激和眼部刺激等。该方法经过MCC等标准验证可靠性较高,而且该工具还提供了一个基于Shapley AdditiveexPlanations(SHAP)方法的用于解释毒性来源基团的展示功能。
背景
机器学习(ML)在毒理学中的目标是开发新的基于人工智能(AI)的计算模型,以预测化学物质的毒性特性。用于计算毒理学的机器学习模型有助于减少体外和体内研究所需的大量成本和长时间,在允许处理大量数据的同时也避开了诸多的伦理限制。在过去的几年里,该领域已经进行了许多努力来收集和评估化学物质的多种性质和毒性的数据;此外,各种不同类型的公司之间也已经应用和分享了新的指导方针,以规范和标准化化学化合物的使用、管理和安全协议。
当下网络工具正在成为计算毒理学预测的基本来源,这是因为它们可以免费获取,而且可以由没有经验的用户使用。因此,创建一个用于毒性预测的网络工具是一种吸引人且必要的工作,以减少动物试验的数量并加速新药的开发。学术界和公司的合作导致了创建不同的开源平台,以便快速和容易地分享关于小分子毒性特性的数据。VEGAHub是关于毒性预测模型和参考数据集的最全面的在线平台之一。VEGAHub提供了各种免费的软件,其中VEGA QSAR是主要的一个,它收集了文献中报告的许多不同的计算方法,并在其中实现,以预测化学物质的毒性特性。
在这个背景下,科学家们最近开发了能够评估小分子的潜在突变性、致癌性、雌激素活性和肝毒性的机器学习模型。所有的模型都使用来自VEGA QSAR的训练和测试集数据进行训练和评估,VEGA QSAR被用作我们的模型性能评估的参考软件。基于多个由不同机器学习模型生成的预测的组合的共识策略,被证明可以提高预测的可靠性,并超过所有参考模型。因此,这种共识的机器学习方法被实现在VenomPred平台中,这是一个免费的毒性预测网络工具,能够快速生成小分子的突变性、致癌性、雌激素活性和肝毒性效应的概率,这些小分子来自于需要分析的化合物的SMILES字符串。
这里报告了VenomPred2.0平台的开发,这是原始网络工具的一个显著升级。具体来说:a) 我们通过包含更广泛的机器学习模型组合,改进了共识机器学习策略,从而进一步提高了所有预测的性能和可靠性;b) 我们生成并包含了用于预测四个新的毒性终点的共识机器学习模型,即雄激素活性、皮肤刺激性、眼睛刺激性/腐蚀性和急性口服毒性;c) 我们实现了SHAP方法,以识别强贡献的毒理学预测的特征,以便推导出结构性毒性元素。
雄激素活性(不希望的雄激素效应)的预测在药物开发的早期阶段成为了一个关键点,以减少在后期人类临床试验中失败的风险,因为已知有多种异物质会与雄激素受体相互作用,破坏正常的内分泌功能,从而导致严重的疾病,如前列腺癌、不育、雄激素不敏感综合症。开发能够有效且可靠地进行此类毒性评估的计算机模型将是十分必要的,以试图减少对体内实验的需求。最后,使机器学习预测更透明、人类可解释、并且整个科学社区容易理解,正在成为需要特别关注的热门话题。事实上,具有复杂架构的机器学习模型可能会达到高预测性能,但以结果可解释性有限为代价,这就产生了需要在预测性和可解释性之间找到平衡,以生成强大且有用的计算机方法。在这个背景下,正在努力发展可解释的人工智能(XAI)策略,以缩小计算和实验化学家以及生物化学家之间的差距,并最大化计算机分析在药物合成规划和安全性提高中的盈利能力。在可以应用于追求这个目标的各种策略中,一个已经建立的方法是基于SHAP方法,该方法仍然被用于确定输入数据特征(通常是分子描述符)对机器学习预测的相关性和影响。
然而,这种方法提供的可解释性强烈依赖于输入特征的性质。这个方面可能产生一个强烈的限制,特别是在药物发现和毒理学领域,其中机器学习模型可能会使用复杂的分子描述符进行训练,这些描述符往往缺乏清晰易懂且易于解释的与化合物的结构特征的连接。因此,简单地在基于分子描述符的机器学习模型中实现基于SHAP的分析,这使得评估单个描述符对预测的影响成为可能,产生的信息对于化学信息学肯定是有用的,但对于寻求明确决策过程(如合成规划或化合物选择)指导的药物化学家和生物化学家的相关性较差,这些决策过程基于计算机毒理学评估。对于科学社区实际有用的是能够从结构的角度解释和解密机器学习模型的预测,直接可视化分析的分子中哪些部分可能具有毒性/不希望的特性。因此,我们最近观察到了一种趋势,即生成基于更易于解释的分子表示的预测模型,如亚结构分子指纹,以及开发能够解码原子和非原子归因的方法,直接在分子结构上表示它们,以直接和容易地解释预测。
受这些最近和有价值的方法的启发,人们开发了一个可靠的基于SHAP的可解释性协议,具有自动归因到结构解码(反向映射),能够直接识别对机器学习预测影响最大的特定分子片段和部分,因此负责小分子的潜在毒理效应。我们的方法确保了对计算机预测的清晰解释,因此可以被整个科学社区轻松利用,不仅可以有助于理解特定的结构-毒性关系,而且还可以为设计更安全的化合物和进一步限制体内实验的需求提供无价的帮助。所有这些功能现在都可以在VenomPred 2.0上找到,并且可以免费获取,地址:
http://www.mmvsl.it/wp/venompred2
训练方法
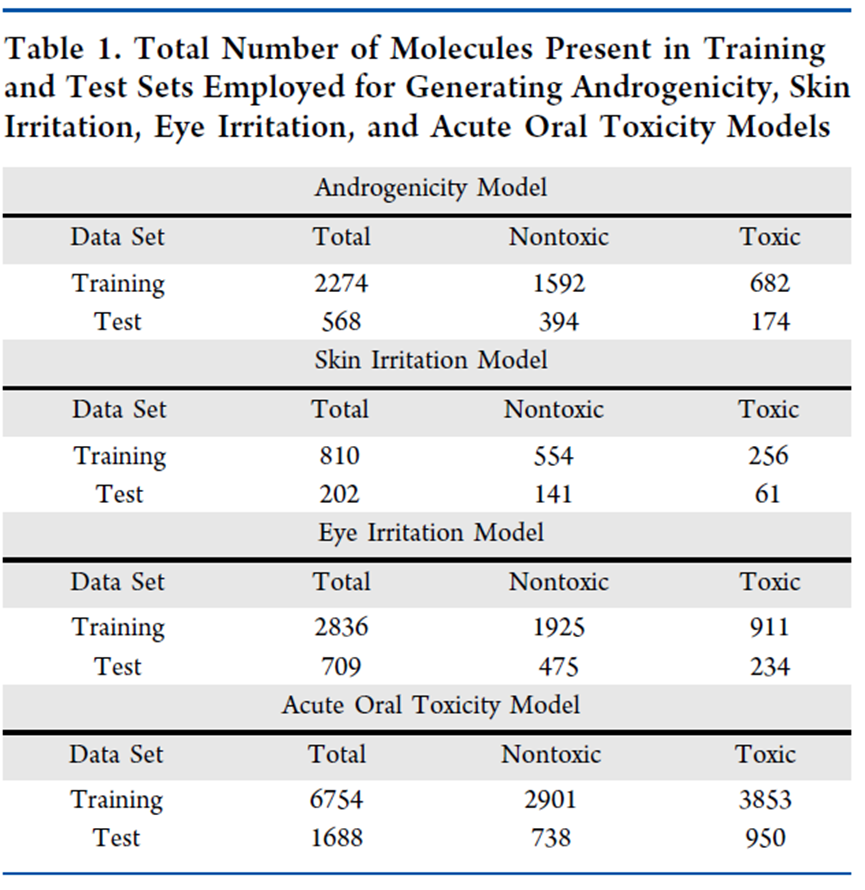
VenomPred 2.0是训练数据来源于ToxCast/Tox21和ChEMBL等多种数据库,对于每个预测项目,首先处理初始数据集以删除不一致和重复的实例;然后,用标准化的SMILES表示化合物的结构。最后,为每个终点获得的精炼数据集被划分为训练集和测试集,分别包含原始数据集的80%和20%,使用的是随机划分策略。所有开发的机器学习模型用于生成和评估四个新终点的数据集的最终组成如表1所示。对于每个终点获得的训练集和测试集都进行了使用t-分布随机邻域嵌入(t-SNE)算法的维度降低处理,该算法应用于编码为PubChem FPs的化合物。分析表明,每个终点的训练集和测试集化合物正确地叠加并覆盖了可比较的化学空间,从而确认每个测试集正确代表了用于模型开发的相应训练集。
对于每个数据集,都计算了化学化合物的分子表示。具体来说,计算了Morgan、RDKit和PubChem的化学指纹(FPs)。Morgan和RDKit的FPs是使用RDKit python库生成的,而PubChem的FPs是使用PyBioMedpython模块计算的。为预测不同毒性终点生成的机器学习模型是使用四种不同的分类算法开发的:随机森林、支持向量机、k-最近邻居和多层感知器,使用python Scikit-learn库的专用函数生成模型。通过使用3种不同的化学指纹和4种不同的机器学习算法,为每个考虑的终点生成了12种不同的毒性模型。基于网格搜索交叉验证的优化过程被应用到所有生成的模型上,以在训练数据集上调整最佳的超参数设置(关于Scikit-learn库的更多细节报告在支持信息中)。
特别地,网格搜索交叉验证包括将训练集划分为几个子集或折叠,并在这些折叠的不同组合上迭代地训练和评估模型。交叉验证的主要目标是通过详尽地评估所有可能的超参数值组合,为它们每个分配一个分数,来估计模型在未见过的数据上的性能。在这项工作中,使用的评分参数是Matthew的相关系数(MCC)。模型预测的特征贡献是通过遵循Shapley值方法来计算的。SHAP方法最初是为了估计一个合作团队中单个玩家的重要性而引入的。用这种方法,团队成员的影响被评估,考虑到他们对游戏最终结果的个人贡献。Shapley值被证明是一种健壮的方法,可以通过获得一个具有以下公理特征的独特结果来公平和合理地评估每个个体的重要性:局部准确性、一致性和零效应。使用SHAP值来解释机器学习模型的想法基于识别直接与模型结果相关的重要特征。
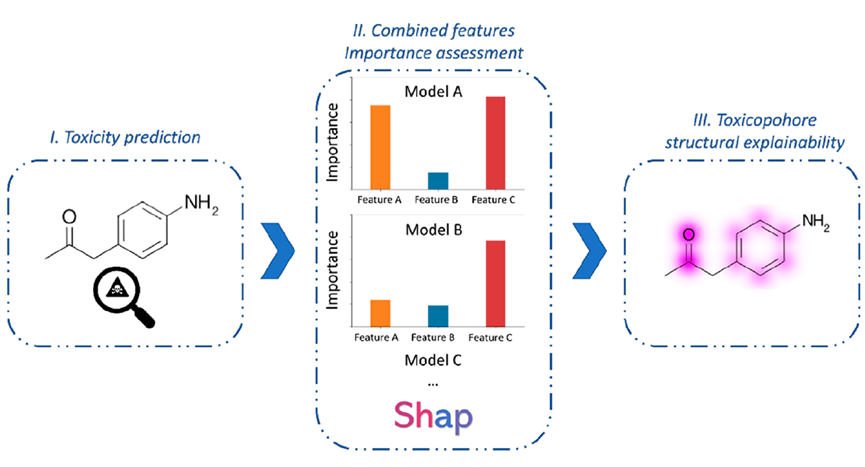
图1. SHAP方法工作流程。
可靠性
对在开发VenomPred中生成的模型进行了性能分析,观察到基于PubChem、RDKit和Morgan指纹的机器学习模型在统计上表现优于使用LINGO和Pharm2D指纹的其他模型。图2显示了基于相同指纹类型的五组模型在Matthew的相关系数(MCC)方面的排名分布。如图所示,基于PubChem、RDKit和Morgan指纹的模型获得的MCC中位排名显著高于其他两组模型。特别地,分布显示大约75%的基于PubChem、RDKit和Morgan指纹的模型在MCC方面的排名不低于11,而大约75%的基于LINGO和Pharm2D指纹的模型排名高于13;因此,基于PubChem、RDKit和Morgan指纹的模型的平均性能似乎明显高于其他模型。
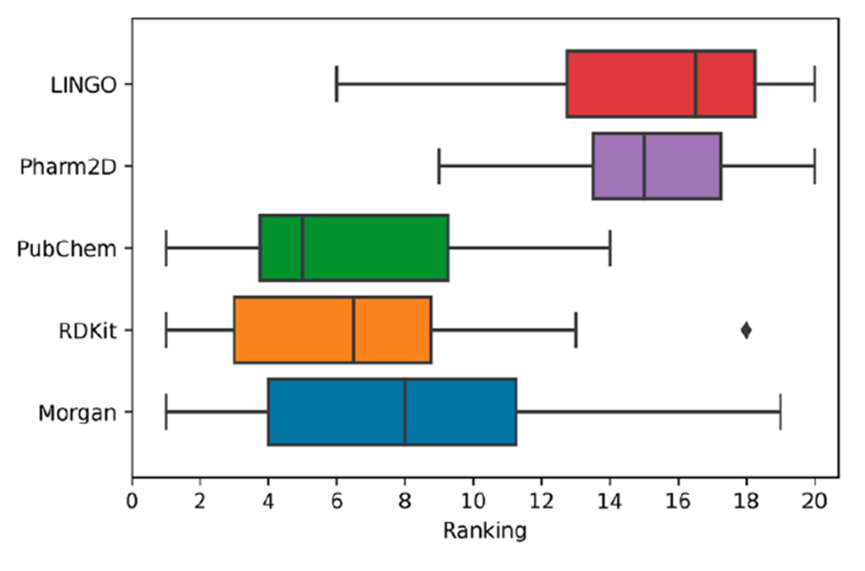
图2. 在VenomPred中基于相同指纹类型先前生成的五组模型在MCC方面的排名分布。
对于每个端点,VenomPred获得了20组不同的预测结果,这使得我们能够对每个生成的模型进行20次不同的性能评估。这些性能是以马修斯相关系数(MCC)来评估的,这是一个可靠的平衡指标,用于评估分类器的性能(详见材料和方法部分)。结果总结在图3中。雄激素活性端点获得了出色的预测性能(超过0.90),而对于其他端点,虽然结果没有那么引人注目,但仍然十分满意。特别是,对于眼睛刺激和急性口服毒性端点,我们得到的MCC值在0.50左右,而皮肤刺激预测模型显示的MCC值在0.4左右。
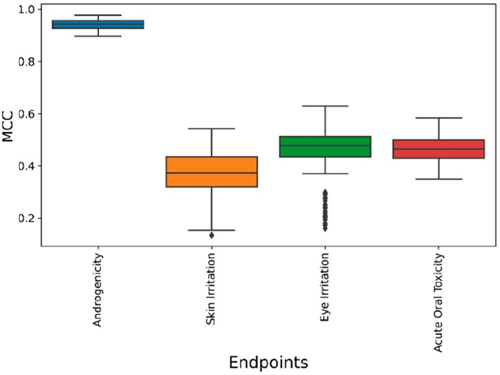
图3. MCC表示的交叉验证结果。
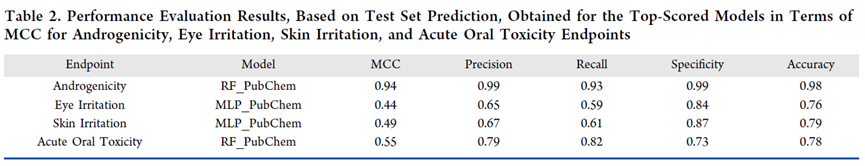
所有为每个新端点开发的模型随后都接受了最终评估,该评估包括预测测试集分子的潜在毒性,这些分子并未用于模型训练。每个端点为此构建的12个模型的性能是以召回率、精确度和特异性来计算的,此外还有准确率和MCC。表2显示了以MCC、精确度、召回率和特异性为标准,每个新端点得到的最高得分模型所达到的性能。实际上,为这个端点生成和优化的所有模型都显示出了出色的结果,MCC值等于或超过0.90。为急性口服毒性端点生成的12个模型显示了平均MCC值约为0.50,最好的模型达到了0.55(见表2),这表明在测试集化合物上进行的毒理预测中,超过75%是正确的,准确率值0.78也证实了这一点。其他两个端点的最佳模型也取得了满意的性能。为眼睛和皮肤刺激的毒理预测生成的模型显示平均MCC值约为0.40,最高可达0.44和0.49,这两个最好的模型都基于MLP算法。然而,他们的性能可靠性得到了精确度值的证实,表明至少有65%被预测为有毒的化合物被正确标记,以及特异性得分超过0.80,表明在预测无害化合物方面具有高可靠性。实际上,这两个模型的准确度值都超过了0.75,证实了测试集预测中超过75%是正确的。
示例
国际癌症研究机构(IARC)将AFB1分类为属于第一组的人类致癌物,因为它通过诱导DNA加合物的形成,促进肝癌的发展。共识致癌性预测正确地将AFB1预测为致癌物,而通过SHAP方法获得的相应特征重要性分析表明,两个五元杂环,特别是二氢呋喃部分(图6A),可能是致癌活性的原因。这些结果与文献和实验数据一致。实际上,AFB1主要在肝脏中被CYP450超家族的氧化酶代谢,产生反应性的8,9-环氧物,存在为两个立体异构体,exo和endo,其中前者被报道为有毒的。这种环氧形式对DNA有高的结合亲和力,形成AFB1-N7-鸟嘌呤DNA加合物,从而导致DNA突变。
对于雌激素活性端点,我们考虑了6-酮雌酮,这是一种强效的17β-羟基固醇脱氢酶类型1(17β-HSD)抑制剂,它是调节雌激素生物可用性的固醇生成酶之一。被确定为负责配体结合到17β-HSD活性位点的共有结构特征之一是酚部分,它与蛋白质的H221和G282残基形成氢键网络。图4B显示,共识预测将6-酮雌酮识别为雌激素化合物,SHAP分析建议酚片段作为分子毒性的责任部分,这与文献研究一致。我们在皮肤刺激端点上验证了SHAP方法,研究了属于丙烯酸衍生物家族的三羟甲基丙烷三丙烯酸酯。这种小分子可以自发聚合或使用催化剂(如紫外光),形成非常耐用的聚合物。丙烯酸单体是强力的致敏化学品,会引起接触性皮炎。在这种情况下,该化合物被预测为皮肤刺激剂,开发的SHAP方法证明了三个丙烯酸基作为潜在的毒性部分,尽管强度略微(图4C)。
为了验证SHAP方法的可靠性,最后考虑的端点是急性口服毒性,其中乙基对硫磷被用作测试结构。这种化合物是一种已知的有机磷杀虫剂,具有一个通常通过抑制乙酰胆碱酯酶来破坏神经系统细胞的有机硫磷基团。实际上,摄入对硫磷后,一个氧化酶将双键硫替换为氧,从而产生对硫磷酮,这比磷酸酯在生物体中更具反应性。这种衍生物作为乙酰胆碱酯酶抑制剂,引起恶心和呕吐、腹痛、腹泻和唾液分泌等典型症状。VenomPred 2.0正确预测了其急性口服毒性,并且基于SHAP分析,硫-磷键被确定为化合物毒性预测的主要贡献者(图4D),这与文献中关于有机硫磷化合物作用机制的报道一致。
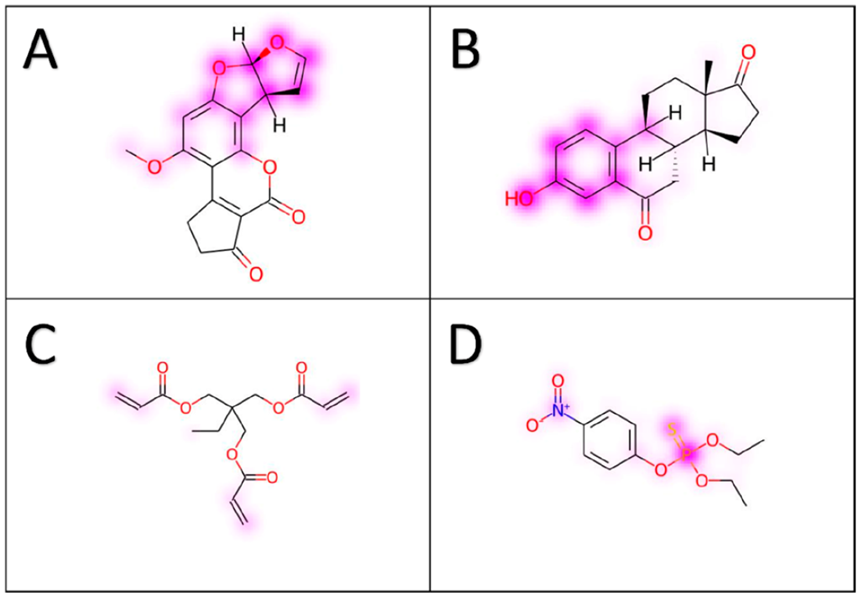
图4.预测案例展示。
获取方式:
网页版:https://www.mmvsl.it/wp/venompred2/
本地版:回复关键词 VenomPred 获取安装包
在Conda环境下,进入安装文件夹根据下面命令即可安装,将化合物的SIMILES存入res.csv文件中,可批量预测分子属性。
#安装
$ conda env create -f enviroment.yml
#预测
$ python VenomPred2_predictions.py -in test.csv -o res.csv参考资料:
Di Stefano M, Galati S, Piazza L, et al. VenomPred 2.0: A Novel In Silico Platform for an Extended and Human Interpretable Toxicological Profiling of Small Molecules[J]. Journal of Chemical Information and Modeling, 2023.
--------- End ---------
